Next-Generation Sequencing
NGS Diagnostik
Zur erweiterten Tumordiagnostik bieten wir Multigenpanels an, welche im Next-Generation Sequencing (NGS) Verfahren untersucht werden. Diese erlauben die gleichzeitige Untersuchung zahlreicher Gene (DNA) und Genprodukte (RNA) auf potentiell tumortreibende Veränderungen sowie Genzahlveränderungen (copy number variations, CNV), Abschätzungen der Mutationslast (tumor mutational burden, TMB) und Mikrosatelliteninstabilität (microsatellite instability, MSI). Die Ergebnisse erlauben einerseits eine detaillierte Charakterisierung des betreffenden Tumortyps [1] und können andererseits auch als Grundlage für personalisierte Therapieentscheidungen dienen.
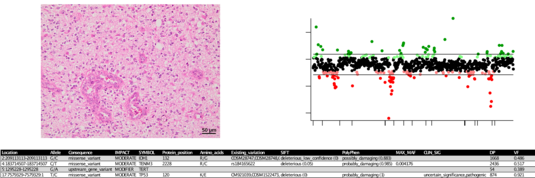
Abbildung: NGS Diagnostik. (A) Repräsentatives histologisches Bild des untersuchten Gewebes, hier ein Glioblastom. (B) Manhattan Plot der Genzahlveränderungen der untersuchten Genregionen, welche hier Deletionen im MYB Gen und am langen Arm von Chromosom 19 (19q) zeigen. (C) Ergebnisse der DNA-Sequenzierung. Angezeigt werden nur Gene mit nachgewiesenen pathologischen Veränderungen. MAX_MAF…maximale bekannte Allelfrequenz in der Normalbevölkerung; DP…Lesetiefe (depth); VF…Variantfrequenz
Epigenetische Analyse
Seltene Hirntumore, zu denen auch viele kindliche Tumore zählen [2], lassen sich mit den Routinemethoden der Histologie, Immunhistologie und konventionellen Molekulardiagnostik oft nicht genau klassifizieren. Ein am DKFZ in Heidelberg entwickeltes Verfahren kann durch die Auswertung epigenetischer Veränderungen (DNA Methylierung) im Genom des Tumors häufig zu einer Klassifizierung beitragen [3–5].
Methode
Für NGS Analysen verwenden wir, je nach Fragestellung, ein eigens entworfenes Genpanel für Hirntumore oder ein allumfassendes onkologisches Panel (Illumina TrusightTM Oncology 500), welche auf Illumina MiniSeq oder NextSeq Plattformen sequenziert werden.
Für epigenetische Analysen verwenden wir das Illumnina Infinium® MethylationEPIC Panel und die Auswerteroutine des DKFZ Heidelberg.
Material
Für NGS Analysen oder epigenetische Analysen genügt die Einsendung von in Formalin fixierten und in Paraffin-eingebetteten (FFPE) oder kryo-asservierten repräsentativen Tumorgewebeanteilen. Für die Untersuchung zellfreier DNA aus Liquor nehmen Sie bitte mit uns Kontakt auf, bevor Sie Proben entnehmen.
NGS Analysen können bei entsprechender Fragestellung an hirneigenen Tumoren, mesenchymalen Tumoren (z.B. Meningeome oder Sarkome) und an Metastasen durchgeführt werden.
Epigenetische Analysen zu diagnostischen Zwecken können nur an hirneigenen Tumoren durchgeführt werden, da für andere Tumorarten noch keine Klassifikatoren vorliegen.
Literatur
1. Louis DN, Perry A, Reifenberger G, von Deimling A, Figarella-Branger D, Cavenee WK, et al. The 2016 World Health Organization Classification of Tumors of the Central Nervous System: a summary. Acta Neuropathologica. 2016;131: 803–820. doi:10.1007/s00401-016-1545-1
2. Sturm D, Orr BA, Toprak UH, Hovestadt V, Jones DTW, Capper D, et al. New Brain Tumor Entities Emerge from Molecular Classification of CNS-PNETs. Cell. 2016;164: 1060–1072. doi:10.1016/j.cell.2016.01.015
3. Capper D, Stichel D, Sahm F, Jones DTW, Schrimpf D, Sill M, et al. Practical implementation of DNA methylation and copy-number-based CNS tumor diagnostics: the Heidelberg experience. Acta Neuropathologica. 2018; doi:10.1007/s00401-018-1879-y
4. Capper D, Jones DTW, Sill M, Hovestadt V, Schrimpf D, Sturm D, et al. DNA methylation-based classification of central nervous system tumours. Nature. 2018;555: 469–474. doi:10.1038/nature26000
5. Jaunmuktane Z, Capper D, Jones DTW, Schrimpf D, Sill M, Dutt M, et al. Methylation array profiling of adult brain tumours: diagnostic outcomes in a large, single centre. Acta Neuropathologica Communications. 2019;7. doi:10.1186/s40478-019-0668-8

